
β-Faltblatt
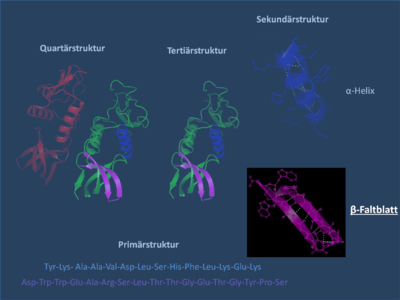
Als β-Faltblatt (engl. β-pleated sheet oder β-sheet) wird in der Biochemie ein häufiges Sekundärstrukturelement eines Proteins bezeichnet. Unter der Sekundärstruktur eines Proteins wird die räumliche Struktur der Aminosäurekette ohne Berücksichtigung der Seitengruppen verstanden. Die Sekundärstruktur eines Proteins geht aus dessen Primärstruktur (Aminosäuresequenz) hervor. Übergeordnete Strukturebenen sind die Tertiärstruktur und die Quartärstruktur. Die dreidimensionale Struktur eines Proteins ist entscheidend für dessen selektive Funktion (siehe Proteinstruktur).
Geschichte
Ende der 1930er Jahre begann William Astbury Kristallstrukturanalysen an kristallinen Peptiden durchzuführen. Dabei wurde festgestellt, dass sich bestimmte räumliche Merkmale regelmäßig wiederholen, bei denen Wasserstoffbrücken innerhalb des Moleküls vermutet wurden. Ihm war jedoch die Planarität der Peptidbindung noch nicht bekannt. Die häufigsten räumlichen Strukturen wurden später α-Helix und β-Faltblatt genannt. Linus Pauling, Robert Brainard Corey und Herman Branson schlugen 1951 ein Modell des β-Faltblatts vor. Das β in „β-Faltblatt“ enthält keine wissenschaftliche Aussage, sondern bringt nur zum Ausdruck, dass das β-Faltblatt nach der α-Helix gefunden wurde. Der Name Faltblatt resultiert aus der dreidimensionalen Struktur des β-Faltblatts, dessen „Zickzack-Form“ einem regelmäßig gefalteten Papierblatt ähnelt.
Struktur

Illustration der Wasserstoffbrückenbindungen, repräsentiert durch gestrichelte Linien bei einem
antiparallelen β-Faltblatt. Sauerstoffatome rot eingefärbt,
Stickstoff Atome
blau.
|

Illustration der Wasserstoffbrückenbindungen, repräsentiert durch gestrichelte Linien bei einem
parallelen β-Faltblatt. Sauerstoffatome rot eingefärbt,
Stickstoff Atome
blau.
|
Aufgrund der Seitenketten sind β-Faltblätter nicht flach, sondern ziehharmonikaähnlich geriffelt (engl. pleated). Die Polypeptidketten eines Faltblattes werden als β-Stränge bezeichnet. Diese Stränge können auf unterschiedliche Weise miteinander verknüpft sein:
- Antiparallele Verknüpfung (gegensinnig): Die Carbonyl- und Aminogruppen eines Strangs sind jeweils über Wasserstoffbrückenbindungen mit der Amino- und Carbonylgruppe des anderen Strangs verbunden
- Parallele Verknüpfung (gleichsinnig): Die NH-Gruppe des einen Strangs ist mit der CO-Gruppe des anderen Strangs über eine Wasserstoffbrücke verknüpft. Die Carbonylgruppe hingegen bildet eine Wasserstoffbrückenbindung mit der Aminogruppe aus, die zwei Reste entfernt vom jeweiligen Partner auf dem anderen Strang liegt. Durch diese Verbindungsweise ist jede Aminosäure des einen Strangs – mit Ausnahme der Aminosäuren an den Enden – mit zwei Aminosäuren des anderen Stranges verbunden. Die Stränge verlaufen in die gleiche Richtung, sodass die einzelnen Atome parallel zueinander angeordnet sind.
- Gemischte Verknüpfung: Bei der gemischten Verknüpfung bestehen sowohl parallele als auch antiparallele Verknüpfungen.

Die dreidimensionale Darstellung erinnert an ein gefaltetes Blatt, wobei die Peptidgruppen in den Flächen und die dazwischenliegenden C-Atome auf den Kanten eines mehrfach gefalteten Blattes liegen. Die Peptidbindungen mehrerer Ketten treten miteinander in Wechselwirkung und bilden Wasserstoffbrückenbindungen entlang des Polypeptidrückgrats aus. Diese treten in Zweierpaaren im Abstand von 7,0 Å auf und sorgen für die Stabilität der Struktur. Der Abstand zwischen vicinalen Aminosäuren ist beim Faltblatt mit 0,35 nm deutlich größer als bei der α-Helix mit 0,15 nm.
Allerdings liegen die Seitengruppen beim β-Faltblatt sehr dicht beieinander, sodass sperrige oder gleich geladene Reste die Anordnung stören. Größere Faltblatt-Strukturbereiche kommen nur dann zustande, wenn die Reste relativ klein sind. So bestehen die Proteinketten der Naturseide, die ausschließlich in der Faltblattstruktur angeordnet sind, zu 86 Prozent aus Glycin, Alanin und Serin, also Aminosäuren mit kleinen Resten. Ein weiteres Beispiel für Proteine mit überwiegender Faltblattstruktur sind Immunglobuline. Liegen überwiegend große Aminosäure-Reste vor, ordnet sich die Polypeptidkette bevorzugt als α-Helix an.
Darstellung

- Bänderdiagramm: In Proteinstrukturen werden β-Faltblätter oft als Pfeile dargestellt. Ein Pfeil entspricht einem Strang des Faltblattes. Mehrere Pfeile, über Turns oder Loops verknüpfte, Pfeile bilden die Faltblattstruktur. Die Pfeile zeigen dabei in Richtung des Carboxyl-Terminus, um die Struktur des Faltblattes wiederzugeben.
- Kalottendarstellung und Kugelstabmodell: Oft werden bei der Darstellung die Wasserstoffatome weggelassen, da sich die Positionen dieser Atome nur schwer bestimmen lassen. Zudem würde unter der Darstellung der Wasserstoffatome die Übersichtlichkeit leiden.
- Rückgratmodell: Es werden meist nur die α-C-Atome dargestellt, wodurch der Verlauf der Polypeptidkette besser zu sehen ist als beim Kalotten- oder Kugelstabmodell. Elemente der Sekundärstruktur sind dennoch nicht sehr gut erkennbar.
Andere wichtige Strukturelemente
Neben der α-Helix und dem β-Faltblatt existieren weitere Arten von Sekundärstrukturmotiven. Die nicht zu einem Motiv gehörenden Teile der Primärstruktur eines Proteins werden Zufallsschleifen (Random-Coil-Strukturen) genannt. Diese Strukturen sind maßgeblich an der Ausbildung der gesamten Proteinstruktur beteiligt.
Weitere häufig vorkommende Motive sind:
Literatur
- Jeremy M. Berg: Stryer Biochemie. Spektrum Akademie Verlag, München 2007, ISBN 978-3-8274-1800-5
Siehe auch


© biancahoegel.de
Datum der letzten Änderung: Jena, den: 13.10. 2023